HOME
>
特集
>
ゲノム編集ツール CRISPR-Cas9 システム特集
HOME
>
試薬
>
遺伝子工学
>
ゲノミクス
>
ゲノム編集
>
ゲノム編集ツール CRISPR-Cas9 システム特集
HOME
>
受託サービス・特注品
>
ゲノミクス・エピジェネティクス
>
ゲノム編集
>
ゲノム編集ツール CRISPR-Cas9 システム特集
ゲノム編集ツール CRISPR-Cas9 システム特集
掲載日情報:2021/06/15 現在Webページ番号:8508
2020年のノーベル化学賞は、ゲノム編集手法の開発に関する業績により、CRISPR-Cas9法の開発者であるエマニュエル・シャルパンティエ博士(ドイツ、マックス・プランク感染生物学研究所)、ジェニファー・ダウドナ博士(米国、カリフォルニア大学バークレー校)の2名が受賞されました。
フナコシ取り扱いのCRISPR-Cas9システムによるゲノム編集ツールをご紹介します。
ゲノム編集に関する総説(東京大学医科学研究所 真下 知士先生ご執筆)
※記載の内容はご執筆時点のものとなります。
追加しました。
- CRISPR-Cas9システムとは
- CRISPR-Cas9の原理
- Casに関する知見
- ゲノム編集のワークフロー(プロトコル)
- 標的配列(ガイドRNA)のデザイン方法
- フナコシ取り扱いメーカーのご紹介
- CRISPR-Cas9システム製品ラインナップ
CRISPR-Cas9システムとは
CRISPR-Cas9 システムは、ゲノム中で任意の領域を切断できる遺伝子改変ツールです。切断したい標的塩基配列に相補的な配列を含むguide RNA(crRNA:tracrRNA) とDNA 切断酵素Cas9 タンパク質により、ゲノム上の任意の配列を切断します。標的配列のデザインの簡便さや実験手法の容易さから、CRISPRはZFN(Zinc Finger Nuclease)やTALEN(Transcription Activator-Like Effector Nuclease)と並び、注目されるゲノム編集技術です。

追加しました。
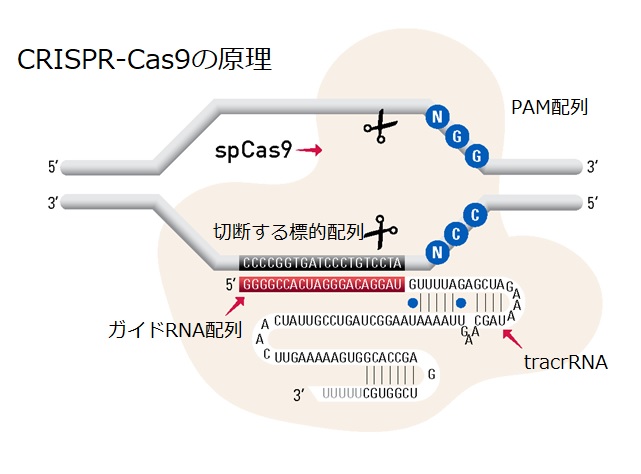
CRISPR-Cas9の原理
2つのコンポーネント:Cas9とガイドRNA
- Cas9ヌクレアーゼ:二本鎖 DNA を切断するハサミの役割
- ガイドRNA(sgRNA または crRNA+tracrRNA):ゲノム上の標的配列を認識する役割
ゲノム編集の流れ
- Cas9 ヌクレアーゼとガイド RNA が複合体を形成
- Cas9/ガイド RNA 複合体がゲノム DNA をスキャンし、ガイド RNA と相同な配列を認識
- PAM 配列上流の DNA に二本鎖切断(DSB;Double Strand Break)を誘導(ガイド RNA の相同配列が PAM 近傍にある時のみ切断)
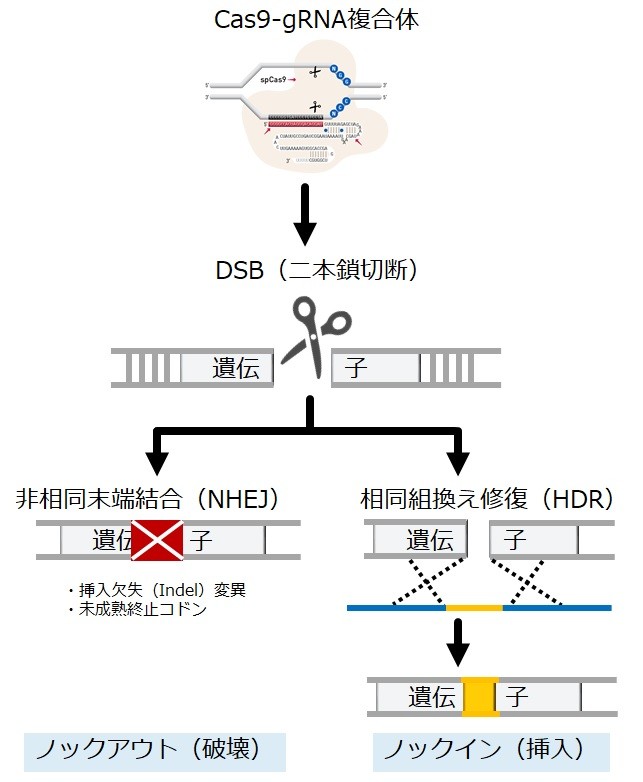
- 二本鎖切断の後、DNA が修復される際の機構(NHEJ、HDR)を利用して遺伝子の改変を行う
●非相同末端結合(NHEJ;Non-homologous end-joining)
塩基のランダムな欠失/挿入(indels)によるフレームシフトを起こす。
⇒ノックアウト
●相同組換え修復(HDR;Homology-directed repair)
切断付近の配列と相同な配列を持つドナー DNA を一緒に導入することで、ゲノム DNA との間に組換えが起き、任意の配列を挿入することができる。
⇒ノックイン or ノックアウト
追加しました。
Casに関する知見
2017年、J. ダウドナ博士のグループはCasX、CasY の発見を報告していました*1が、2019年、CasXはCas9やCas12aとは異なるメカニズムで機能する3つ目のプラットフォームであり、大腸菌やヒト細胞でのゲノム編集が可能であることが示されました*2。
| Cas9 | ・ ヒトによく見られる細菌に由来(spCas9:化膿レンサ球菌、SaCas9:黄色ブドウ球菌) ・切断箇所は平滑末端となる |
|---|---|
| Cas12a(Cpf1) | ・切断にcrRNAが必要だがtracrRNAは必要としない ・切断箇所は突出末端(付着末端)となる |
| Cas13a(C2c2) | ・RNA誘導 RNase ・DNAは切断しない ・ dCas13にRNA置換酵素を結合することでRNA編集が可能 |
| CasX | ・Cas9よりサイズがおよそ40%小さい ・人体には見られない細菌由来 ・ Cas9/Cas12aと共通祖先を共有しない(収斂進化) |
●Casの亜種
- dCas9(deadCas9):ヌクレアーゼ活性欠損型Cas9。DNA配列に結合するが、切断しない
- nCas9(nickase Cas9):一本鎖ニックを導入
- xCas9:広いPAM適合と高いDNA特異性を持つ
●dCasの応用
- 転写活性化因子を融合:CRISPRa(activation)
- RNAポリメラーゼを立体的にブロックまたは転写抑制因子を融合:CRISPRi(interference)
- DNA(脱)メチル化酵素を結合:エピゲノム編集
■ 参考文献
*1 Burstein D., et. al.,“ New CRISPR-Cas systems from uncultivated microbes.” Nature 542: 237~241(2017)[PMID: 28005056]
*2 Liu J.J., et. al.,“ CasX enzymes comprise a distinct family of RNA-guided genome editors.” Nature 566: 218~223(2019)[PMID:
30718774]
*3 Yao, Ruilian, et al.“ CRISPR-Cas9/Cas12a biotechnology and application in bacteria.” Synthetic and Systems Biotechnology(2018)
追加しました。
ゲノム編集のワークフロー(プロトコル)
切断したいDNA配列に対する相補的な配列を含むguide RNA、およびその相補鎖を設計し、それらの断片をCRISPRベクターにクローニングします。作成したCRISPR-Cas9発現プラスミドを目的細胞へトランスフェクションすると、CRISPR-Cas9システムにより標的領域が切断されます。
![]() デザイン
デザイン
ノックアウト、ノックイン、編集、タグの挿入などを行う標的配列に対して、guide RNAと相同組換えドナープラスミド(HR donor plasmid)を設計する。
![]() プラスミドの構築
プラスミドの構築
guide RNAをall-in-one Cas9 vectorにクローニングする。5’および 3’側のホモロジーアームをHR donor plasmidにクローニングする。遺伝子をノックインする場合は目的遺伝子もクローニングする。
![]() 細胞への導入
細胞への導入
Cas9, guide RNA(およびドナーDNA)を目的の細胞へトランスフェクション(トランスダクション)またはインジェクションにより導入する。
![]() 細胞の選択
細胞の選択
蛍光レポーターの発現確認(フローサイトメトリー)や抗生物質選択により、導入細胞を選択、濃縮する。
![]() バリデーション
バリデーション
片方または両方のアレルがゲノム編集された細胞について、標的遺伝子の配列を確認する。
PCR、DNA シークエンシング、ミスマッチ検出、ウェスタンブロッティング、表現型の解析など
追加しました。
標的配列(ガイドRNA)のデザイン方法
標的DNA配列は、末端に「GG」が配置されている領域を選択し、NGG の上流20 塩基で設計します。切断したい標的配列を含むgRNA およびその相補鎖をオリゴ合成等で作成します。
| 標的領域 | 5’ NNNNNNNNNNNNNNNNNNNNNGG 3’ |
| デザインする配列の例 | 5’TGTATGAGACCACNNNNNNNNNNNNNNNNNNNN 3’ 3’ACTCTGGTGNNNNNNNNNNNNNNNNNNNNCAAA 5’ |
※ ベクターにより、標的配列の両端に付加する配列は変わります。
※ ウェブ上で公開されているguide RNA 設計用のソフトウェア⇒ CHOPCHOP, CRISPRdirect
変異型 Cas9、Cas9 Nickase(Cas9(D10))について
Cas9 タンパク質に D10A アミノ酸変異が生じると、ヌクレアーゼ活性が不活性化し、ニッカーゼ活性を持つようになります。*4。Cas9(D10A)は、野生型Cas9 のようにDNA 二本鎖を切断せず、一本鎖のみを切断しニックを入れます。そのため相同組換えによるゲノム編集において、二本鎖切断により引き起こされるDNA 修復機構NHEJ(非相同末端再結合)が起こらず、望ましくない領域での遺伝子欠失・挿入やオフターゲット効果を抑制することができます* 5。また、最近では2種類のguide RNA を用いて2か所にニックを入れることで、二本鎖切断によるノックアウトで細胞株におけるオフターゲット効果を50 ~ 1,500 倍まで減少できた例が報告されています* 6。
Cas9 のDouble Mutant (D10A, H840A) はヌクレアーゼ活性およびNickase 活性を有していませんが、guide RNA 標的配列への結合能を有しています。オフターゲット効果の少ない転写リプレッサーとして機能すると考えられており* 7、部位特異的な転写制御の研究への応用が期待されています。
■ 参考文献
*4 Jinek, M., et al., Science, 17, 337(6096), 816 ~ 821 (2012).
*5 Cong, L., et al., Science, 15, 339(6121), 819 ~ 823 (2013).
*6 Ran, F. A., et al., Cell, 12, 154(6), 1380 ~ 1389 (2013).
*7 Qi LS., et al., Cell, 152 (5): 1173-83 (2013).
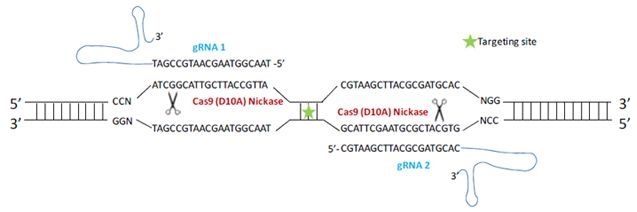
標的部位のセンス鎖に対するguide RNA 1 とアンチセンス鎖に対するguide RNA 2 を作製し、Cas9 (D10A) を用いて2か所でニックを入れ、オフターゲット効果を抑制する。
追加しました。
フナコシ取り扱いメーカーのご紹介
追加しました。
CRISPR-Cas9システム製品ラインナップ
Cas9プラスミド/mRNA/タンパク質/レンチベクター
任意のガイドRNAを組み込めるCas9ベクター
- Cas9とガイドRNAを同時発現できるプラスミドベクター SBI
- Cas9とガイドRNAを同時発現できるレンチウイルスベクター DHA
- Cas9とガイドRNAを同時発現できるレンチウイルスベクター SBI
ガイドRNA
- nCas9、APOBEC、化学合成sgRNAによる二本鎖切断を伴わないゲノム編集システム DHA NEW!!
- デザイン済みのガイドRNA発現用レンチウイルスベクター(sgRNA) DHA
- デザイン済み化学合成ガイドRNA(crRNA/tracrRNA) DHA
- デザイン済み化学合成ガイドRNA(sgRNA) DHA
- EasyEdit sgRNA / SafeEdit sgRNA合成受託サービス GSC NEW!!
- NHEJを用い高効率導入ができるデザイン済みガイドRNA発現用プラスミド ORI NEW!!
- デザイン済みガイドRNA発現用プラスミド ORI
- in vitro転写(IVT)ガイドRNA作製キット SBI
- ガイドRNAのクローニングキット SBI
- 網羅的なノックアウトスクリーニングに有用なガイドRNAライブラリー DHA
- Horizon Discovery ガイドRNAの設計オンラインツール DHA
- お手持ちのDNAから簡便にsgRNAライブラリーを調製できるキット PIB NEW!!
■ガイドRNA
sgRNA(single guide RNA キメラ単鎖 RNA):ゲノム編集効率が高い(Cas9 タンパク質との同時導入)
crRNA:tracrRNA (CRISPR RNA: trans-activating CRISPR RNA ハイブリッド):短鎖 RNA のため取り扱い/コスト有利
ドナーDNA
- GenWand 二本鎖DNA受託作製サービス GSC NEW!!
- CRISPRノックインに有用な一本鎖DNA合成受託サービス GSC
- 人工遺伝子合成受託サービス GSC
- 長鎖一本鎖DNAの調製キット BDL
- ISによる変異がプラスミドに入りにくい!ゲノム編集済みコンピテントセル BDL
- 長鎖一本鎖DNA専用ゲル抽出キット BDL
- ノックアウト/ノックイン/編集用ドナーベクター SBI
- 相同組換え用ドナーベクターPiggyBac SBI
- ノックイン用ドナープラスミド構築キット DHA
- Horizon Discovery ドナーDNAの設計オンラインツール DHA
■HR ドナーベクター
Cas9と組み合わせて使用します。
Cas9単体でも DSB(二本鎖切断)により遺伝子ノックアウトが可能ですが、ノックアウト用の HRドナーベクターを用いることで、相同組換えに成功した細胞を効率よく選択できます。また、Cas9で切断した部位に特定の配列をノックインしたい場合には、その目的配列を含むドナーベクターを一緒にトランスフェクションすると、相同組換え(HDR)によりその配列をノックインすることができます。
細胞への導入
- 細胞毒性が低く抑えられたトランスフェクション試薬 DharmaFECT1~4 DHA
- 細胞毒性が低く抑えられたトランスフェクション試薬 DharmaFECT Duo DHA
- 細胞毒性が低く抑えられたトランスフェクション試薬 DharmaFECT kb DNA DHA
- プラスミドDNAのトランスフェクション試薬 PPU
- Cas9タンパク質、Cas9/ガイドRNA RNP複合体導入用試薬 OZB
- 容量可変型オートインジェクター DRM
- 容量可変型オートインジェクター(エントリーモデル) DRM
ゲノム編集の確認
- ゲノム編集効率の確認キット BDL NEW!!
- ゲノム編集された細胞数の割合を測定できる qPCR用マスターミックス SBI
- Cas9 mRNA発現を確認する:Cas9用プライマーのセット SBI
- Cas9導入クローンのスクリーニング:Cas9タンパク質定量用ELISAキット EPG
- 一塩基違い(SNP)をPCRで検出できるポリメラーゼ MYP
- キャピラリー式電気泳動装置 BOP
- 単一細胞の単離、培養(シング ルセルクローニング)用デバイス ORG
- ノックイン遺伝子の挿入位置を確認する:外来DNAの挿入位置解析受託サービス PGL
受託サービスを利用する
- 遺伝子改変マウス作製 受託サービス TRG
- 遺伝子改変細胞株の作製 受託サービス YNK
- ノックアウト変異植物作製 受託サービス INP
- 大腸菌(E. coli)ゲノム編集サービス GSC NEW!!
CRISPR-Cas9を利用して転写を活性化する
- 標的遺伝子の転写活性化(CRISPRa)ができるレンチウイルスのall-in-oneシステム DHA NEW!!
- 標的遺伝子の転写を活性化させるシステム(CRISPRa) DHA
- SAMシステムを利用して標的遺伝子の転写を活性化するための化学合成tracrRNA DHA
- 標的遺伝子の転写を活性化させるプラスミドベクター(CRISPRa SAM) ORI
CRISPR-Cas9を利用して転写を抑制する
- 標的遺伝子の転写抑制(ノックダウン、CRISPRi)ができるレンチウイルスのall-in-oneシステム DHA NEW!!
- 標的遺伝子の転写を抑制(ノックダウン)させるシステム(CRISPRi) DHA
- 標的遺伝子の転写を抑制(ノックダウン)させるプラスミドベクター(CRISPRi) ORI
追加しました。
おすすめ製品記事
-
BioDynamics Laboratory Inc.(株式会社バイオダイナミクス研究所)
/ 非変性アガロースゲルでのRNA電気泳動が簡単に行えるバッファーとマーカーのセット
DynaMarker® RNA High for Easy Electrophoresis
製品情報は掲載時点のものですが、価格表内の価格については随時最新のものに更新されます。お問い合わせいただくタイミングにより製品情報・価格などは変更されている場合があります。
表示価格に、消費税等は含まれていません。一部価格が予告なく変更される場合がありますので、あらかじめご了承下さい。