NHEJを利用し、幅広い細胞で高効率に遺伝子ノックアウトを行えます KN2.0 Non-homology Mediated CRISPR Gene Knockout Kit
掲載日情報:2023/03/15 現在Webページ番号:68457
追加しました。
- CRISPR/Cas9を用いた遺伝子ノックアウトキットについて
- KN2.0 Kitによる遺伝子ノックアウトの概要
- 特長
- 操作方法概略
- KN2.0 Kitによる遺伝子ノックアウトの検証例
- ウェビナー動画
- KN2.0 KitのFAQ
- 納品物
- 価格
- ご注文方法
- 使用文献について
- 関連した特集記事
CRISPR/Cas9を用いた遺伝子ノックアウトキットについて
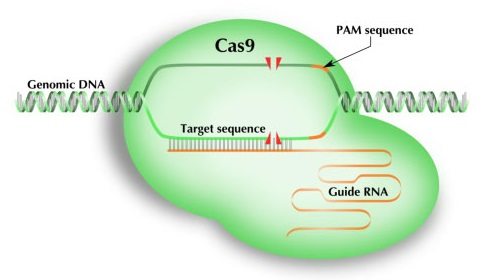
CRISPR/Cas9を用いた遺伝子ノックアウト(Gene Knockout、KO)では、細胞内の修復機構について相同組換え(HDR、Homology Directed Repair)を利用する場合と、非相同末端結合(NHEJ:Non-homologous End Joining)を利用する場合があります。
本キットは、NHEJを利用しており、非分裂細胞を含めた幅広い細胞で、HDRを利用したキットよりも高効率に遺伝子ノックアウトを行えます。
OriGene社の遺伝子ノックアウトキットの比較
| キット名 | HDR Mediated Kit | KN2.0 Non-homology Mediated Kit (本製品) |
|---|---|---|
| gRNA Vector | pCas-Guide | |
| ドナーカセット | Homologous Armが両端に隣接 | Homologous Armなし |
| 細胞スペクトル | 分裂細胞 | 分裂および非分裂細胞 |
| ノックアウト効率 | 中 | 高 |
追加しました。
KN2.0 Kitによる遺伝子ノックアウトの概要
追加しました。
特長
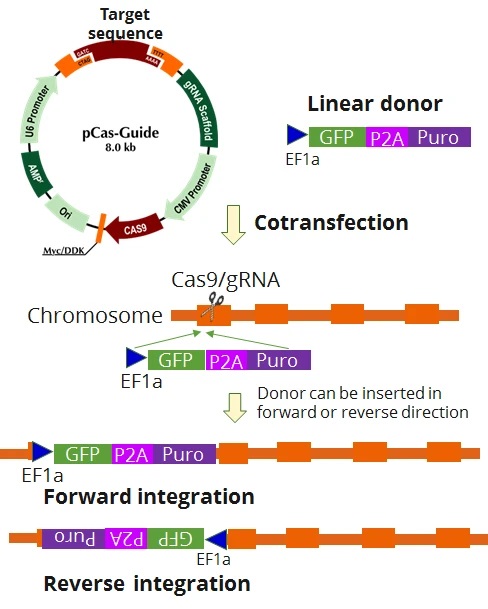
- gRNAベクター2種とドナーDNAをセットにしたReady-to-Useのキットです。
- 分裂細胞、非分裂細胞いずれにおいても、高効率で遺伝子ノックアウトが行えます。
- ドナーDNAに選択マーカー配列が含まれるため、スクリーニング操作が容易です。
- ヒトおよびマウスの幅広い遺伝子についてキットを取りそろえています。
追加しました。
操作方法概略
追加しました。
KN2.0 Kitによる遺伝子ノックアウトの検証例
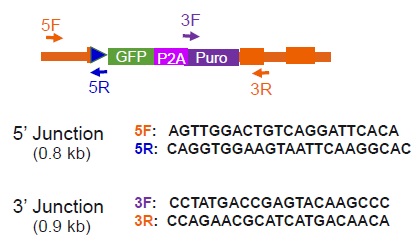
ゲノムPCRによるドナーDNA組込みの確認
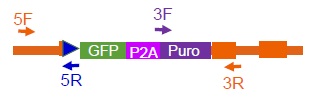
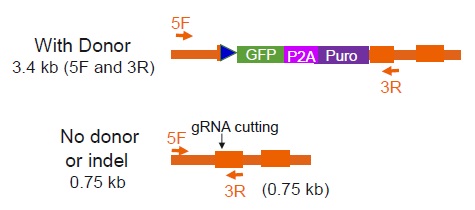
ゲノムPCRのプライマーのデザイン
※ 5Fおよび3Rプライマーは標的遺伝子に特異的な物が必要で、その配列は標的遺伝子のゲノム配列からデザインする必要があります。
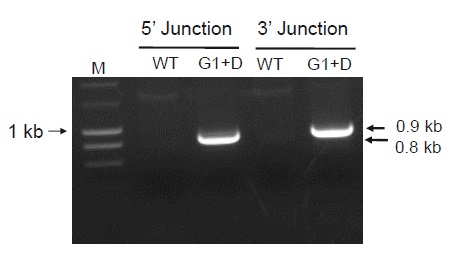
順方向でのゲノム組込みの検証
逆方向でのゲノム組込みの検証
細胞クローンにおけるゲノム組込みの方向の確認
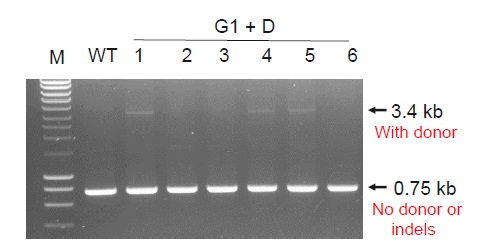
HEK293細胞にgRNA1とドナーDNAをトランスフェクトし、ピューロマイシン選択を行って得られた安定細胞プールから単一細胞コロニー(1~6)を単離した。単離した細胞からゲノムDNAを抽出し、5F/3Rプライマーの組合せでゲノムPCRを行った。WTは未改変のHEK293細胞から抽出したゲノムDNA。
ゲノム編集なしおよびindelが生じた場合は0.75 kbのPCR産物が、ドナーDNA組込みが生じた場合は3.4 kbのPCR産物が得られる。
6つの細胞コロニー中の、1、4、5の3つのクローン(50%)で、ドナーDNAの組込みが生じていることが示された。
シークエンシングによるノックアウトの確認
0.75 kbのPCR産物をシークエンシングして、ノックアウト部位でのindelを確認した(両アレルについて、一方にドナーDNAの組込み、もう一方にindelが生じていることが確認された)。
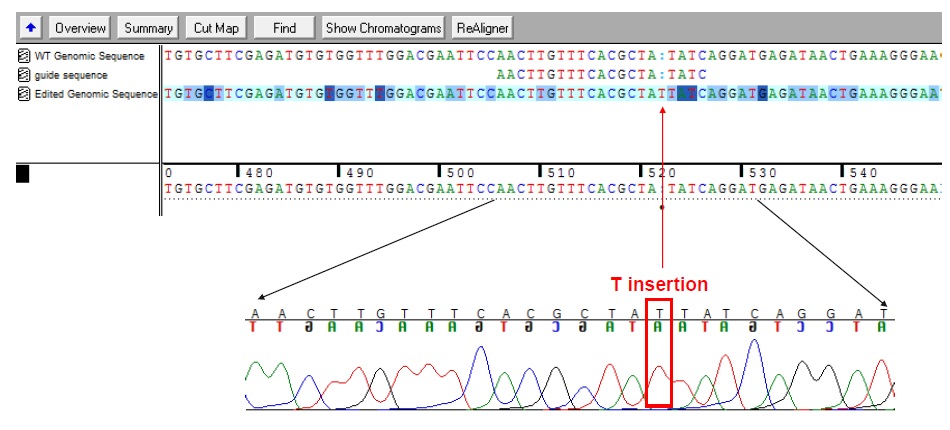
確認例1:ノックアウト部位にT(チミン)の挿入が確認された
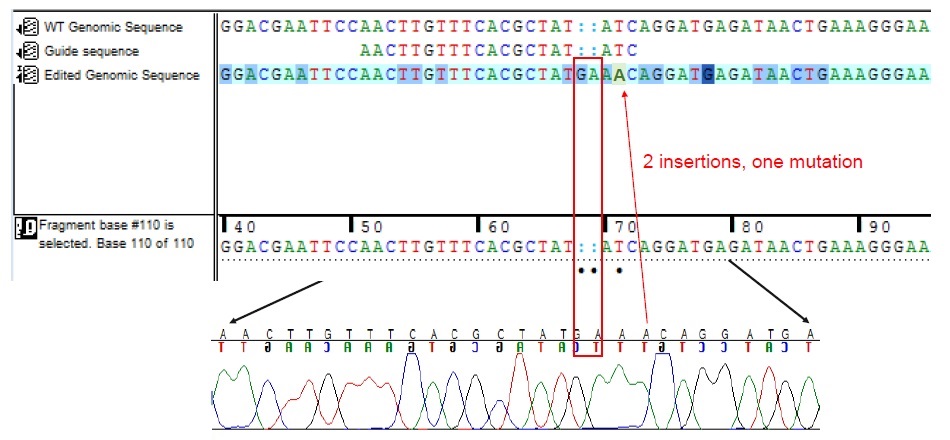
確認例2:ノックアウト部位に2塩基の挿入と1塩基の置換が確認された
MIA PaCa-2細胞中のSHMT1遺伝子のノックアウト
KN2.0 CRISPR Gene Knockout Kitを用いて、MIA PaCa-2(ヒト膵がん由来細胞株)中のSHMT1遺伝子をノックアウトした(ユーザー様提供データ)。
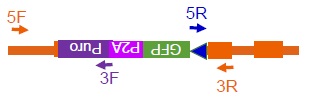
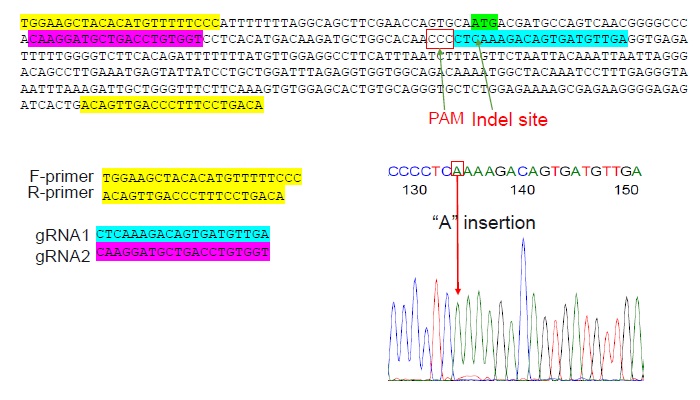
gRNAおよびドナーDNAのデザイン
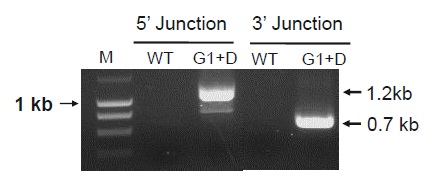
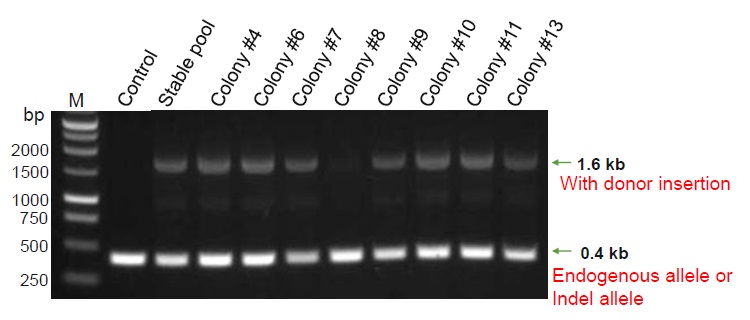
安定細胞プールにおけるゲノムPCR
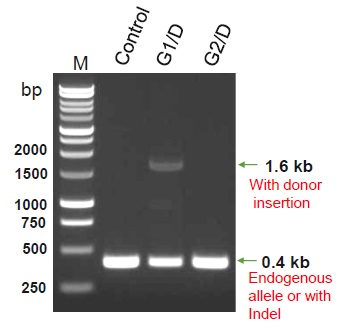
MIA PaCa-2細胞にgRNA(G1またはG2)とドナーDNA(D)をトランスフェクトし、8回継代した後、ピューロマイシン選択を行った。得られた安定細胞プールからゲノムDNAを抽出し、ゲノムPCRを行った。
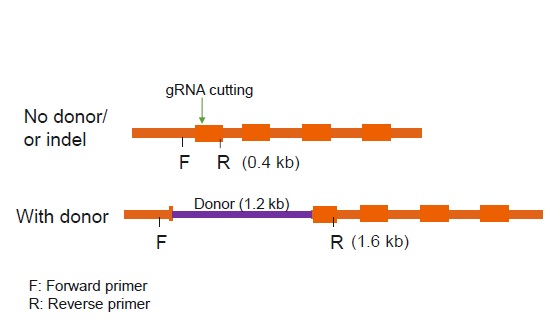
ドナーDNA組込み部位の両隣にPCRプライマーをデザインし、ゲノム編集なしおよびindelが生じた場合は413 bpのPCR産物が、ドナーDNA組込みが生じた場合は1.6 kbのPCR産物が得られる。
コントロールは未改変のMIA PaCa-2細胞から抽出したゲノムDNA。
gRNA1(G1)を用いた安定細胞プールで、ドナーDNAの組込みが生じていることが示された。
単一細胞のコロニーにおけるゲノムPCR
シークエンシングによるノックアウトの確認
0.4 kbのPCR産物をシークエンシングして、ノックアウト部位でのindelを確認したところ、A(アデニン)の挿入が確認された(両アレルについて、一方にドナーDNAの組込み、もう一方にindelが生じていることが確認された)。
ウエスタンブロットによるノックアウトの確認
追加しました。
ウェビナー動画
CRISPR gene knockout webinar to get high biallelic knockout(英語、約50分)
追加しました。
KN2.0 KitのFAQ
Q-1. 標的遺伝子をノックアウトする際には、ドナーDNAは必要ですか?
A-1. ドナーDNAがないと、二本鎖切断は非相同末端結合(NHEJ:non-homologous end joining)によって修復され、予期せぬindel(挿入・欠失)が導入される傾向があります。そのため、標的遺伝子のフレームシフトを引き起こすような挿入・欠失についてスクリーニングをする必要があります。ドナーDNAに薬剤耐性遺伝子やGFP発現遺伝子を組込むことで、下流のスクリーニング手順を大幅に簡略化することができます。
Q-2. KN2.0 CRISPR Gene Knockout Kitによる遺伝子ノックアウトとドナー配列の標的遺伝子への組込みのメカニズムはどのようになっていますか?
A-2. KN2.0 Kitは、CRISPR-Cas9を用いた標的ゲノム編集技術をベースにデザインされています。標的遺伝子に特異的なgRNAがゲノムを切断し、選択カセットを含むドナーDNAが非相同末端結合(NHEJ:non-homologous end joining) を介した修復機構により切断部位に組込まれます。
ドナーDNAは、ゲノム中に順方向・逆方向のいずれかで組込まれるため、ドナーDNAに含まれる選択マーカー(ピューロマイシン耐性遺伝子)とレポーター遺伝子(GFP)を利用してスクリーニングを行います。
本キットを用いた遺伝子ノックアウトでは、多くの場合において1回のゲノム編集で両アレル(biallelic)をノックアウトします。そして、その多くの場合において、一方のアレルにドナーカセットが組込まれ、もう一方のアレルにはindel(挿入・欠失)が生じます。
Q-3. KN2.0 CRISPR Gene Knockout Kitの利点は何ですか?
A-3. 相同組換え(HDR)を介した遺伝子ノックアウト/ノックインは、手法が確立されていますが、HDR効率の低い細胞種や生物種では必ずしも適用できない場合があります。
KN2.0 Kitは、あらゆる細胞種や生物種に対する遺伝子ノックアウトのニーズに対して、普遍的な手段を提供するために特別に設計されており、ノックアウト効率が向上していることを確認済みです。
Q-4. KN2.0 CRISPR Gene Knockout Kitでの使用実績がある細胞は何ですか?
A-4. HeLa、HEK293T、MIA PaCa-2(ヒト膵がん由来細胞株)があります。
Q-5. KN2.0 CRISPR Gene Knockout Kitでピューロマイシン選択を行ったところ、生存細胞がありませんでした。この場合、どのような原因が考えられますか?
A-5. 2つの可能性が考えられます。
まず、その遺伝子が細胞生存に必須の遺伝子であるため、構成的な遺伝子ノックアウトに耐えることができないケースです。この場合は、コンディショナルノックアウトを行うが必要があります。
もう一つは、トランスフェクションの効率が低すぎるケースです。この場合は、トランスフェクションの最適化、またはエレクトロポレーション法などの別のトランスフェクション方法を選択する必要があります。
Q-6. KN2.0 CRISPR Gene Knockout Kitは、トランスジェニック動物モデル作製のための胚マイクロインジェクションに使用できますか?
A-6. KN2.0 Kitはトランスジェニック動物モデルを作製するための胚マイクロインジェクションに、理論上は使用することができます。しかしながら、OriGene社では未検証で、お客様自身での最適化が必要です。
追加しました。
納品物
- gRNA配列が組込まれたpCas-Guide Vector(2種類)
- Linear donor DNA(1種類)
追加しました。
価格
価格についてはお問合せ下さい。
追加しました。
ご注文方法

OriGene社ウェブサイトにアクセスし、上部にある検索ボックスに遺伝子やAccession No.など、および「non-homology」を入力して下さい(その際に半角スペースを開けて下さい)。

「KN2.0」および「non-homology mediated」と記載されている製品が該当となります。
商品コードをお知らせ下さい。
追加しました。
使用文献について
OriGene社CRISPR Gene Knockout Kitの使用文献については、以下の2種類の方法でご確認いただくことができます。
※ 厳選されたOriGene社製品の、代表的な使用文献のみを掲載しています。
① 使用文献一覧
OriGene社CRISPR Gene Knockout Kitの使用文献一覧はこちらからご確認いただくことができます。
※ 表示される文献は、KN2.0 Non-homology Mediated Kit(本キット)とHDR Mediated Kitの使用文献の合計となります。
② 各製品の使用文献
OriGene社ウェブサイトのSearchボックスで目的キットの検索を行い、検索結果を表示させると、文献掲載実績がある製品の場合、使用文献数が表示されます(下図参照)。
さらに製品名をクリックし、製品詳細のページへ移行すると、文献情報をご確認いただけます。

使用文献の例
使用した製品:ABCB5, Human, CRISPR KN2.0 Kit(#KN415604)
- Milosevic, V., et al., Int. J. Cancer, 146 (1), 192 (2020). [PMID: 31107974]
追加しました。
関連した特集記事

追加しました。
製品情報は掲載時点のものですが、価格表内の価格については随時最新のものに更新されます。お問い合わせいただくタイミングにより製品情報・価格などは変更されている場合があります。
表示価格に、消費税等は含まれていません。一部価格が予告なく変更される場合がありますので、あらかじめご了承下さい。