HOME
>
試薬
>
遺伝子工学
>
ゲノミクス
>
ゲノム編集
>
CRISPY Master Mix for qPCR-based Gene Editing Quantitation
HOME
>
試薬
>
遺伝子工学
>
PCR・シークエンシング
>
定量PCR/リアルタイムPCR
>
CRISPY Master Mix for qPCR-based Gene Editing Quantitation
CRISPR/Cas9によるゲノム編集効率を簡単に測定できます! CRISPY Master Mix for qPCR-based Gene Editing Quantitation
掲載日情報:2020/12/04 現在Webページ番号:69912
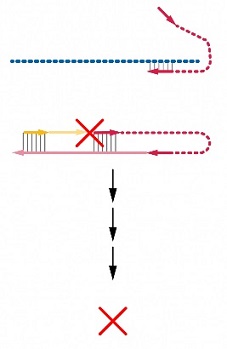
CRISPYアッセイを用いて、CRISPR/Cas9でゲノム編集された細胞数の割合を迅速、高感度かつ簡単に測定できるqPCR用マスターミックスです。スナップバックプライマー(別途用意)、および5'→3'エキソヌクレアーゼと鎖置換活性の両方を欠いたDNAポリメラーゼを用いるCRISPYアッセイは、ミスマッチ検出アッセイに比べてあらゆる点で優れています。
追加しました。
CRISPYアッセイの原理
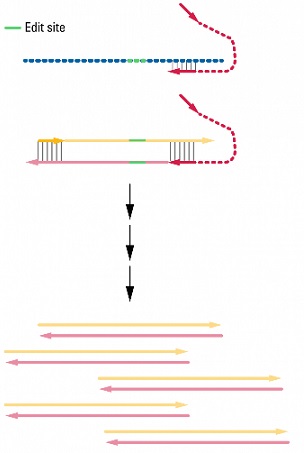
A.プライマーの設計
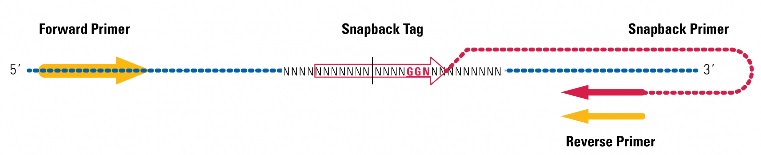
プロトスペーサー隣接モチーフ(PAM, protospacer adjacent motif)配列の先頭塩基(GGN)から3~4塩基上流の塩基を中心とする14~16 nt を選択し、スナップバックタグを設計する。未編集配列と相同なスナップバックタグと、および5'→3'エクソヌクレアーゼ活性と鎖置換活性がないDNAポリメラーゼを用いてqPCRを行う。

追加しました。
特長
- 標的領域における編集効率を測定できます。
- ワンステップのワークフローで、1時間以内に測定結果が得られます。
- DNAの精製は不要です。
- 最低0.5 ngのDNA量で測定が可能です。
- 高感度で、1%という低いゲノム編集成功率の検出が報告されています。
- ゲルイメージングに代わりCt値に基づいて定量を行うため、より信頼性ある結果が得られます。
- 使用回数:200 reactions
※ 測定に必要なプライマーは含まれておりません。別途、本アッセイ専用にスナップバックプライマーを設計いただく必要があります。
追加しました。
使用例
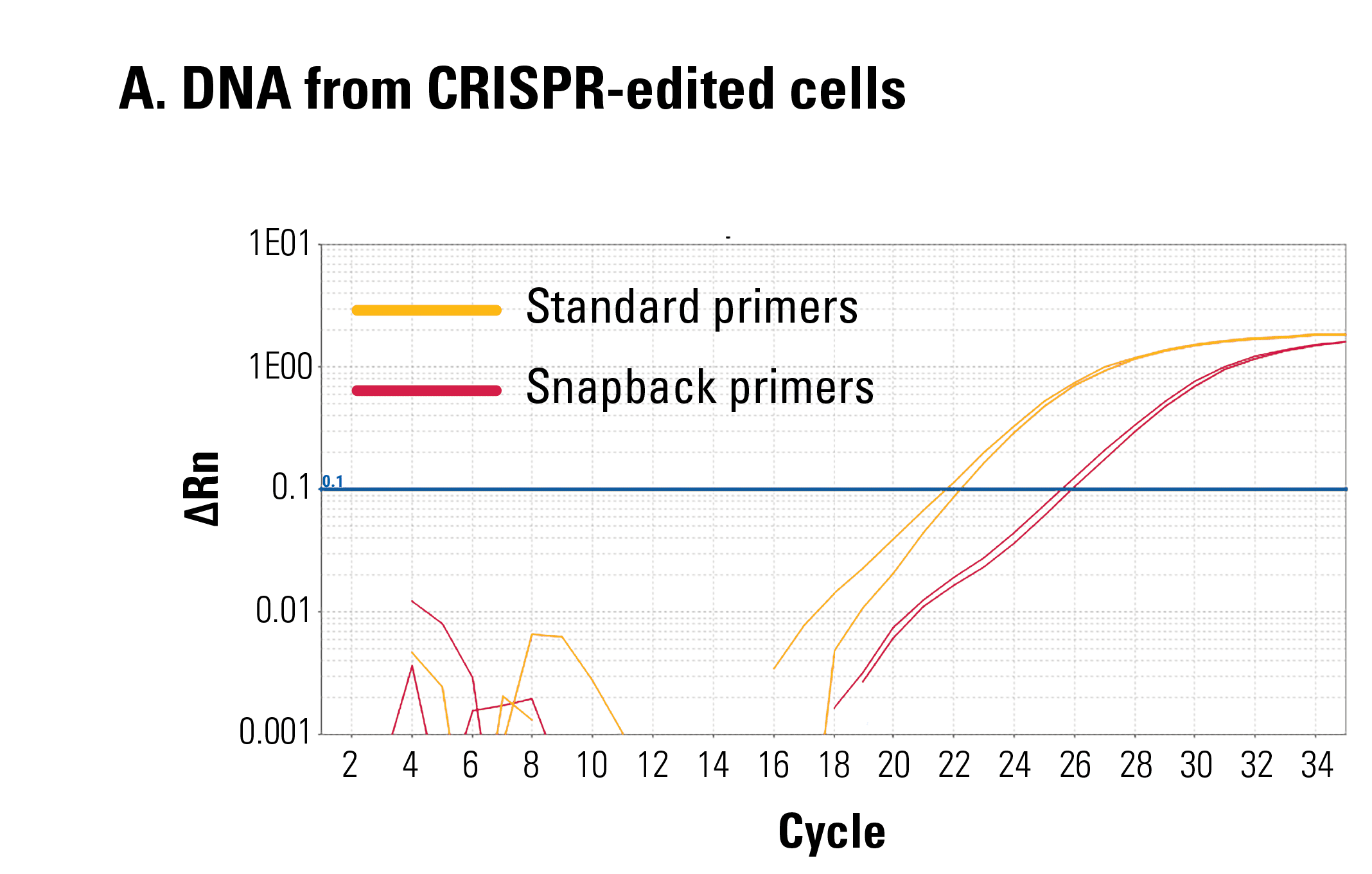
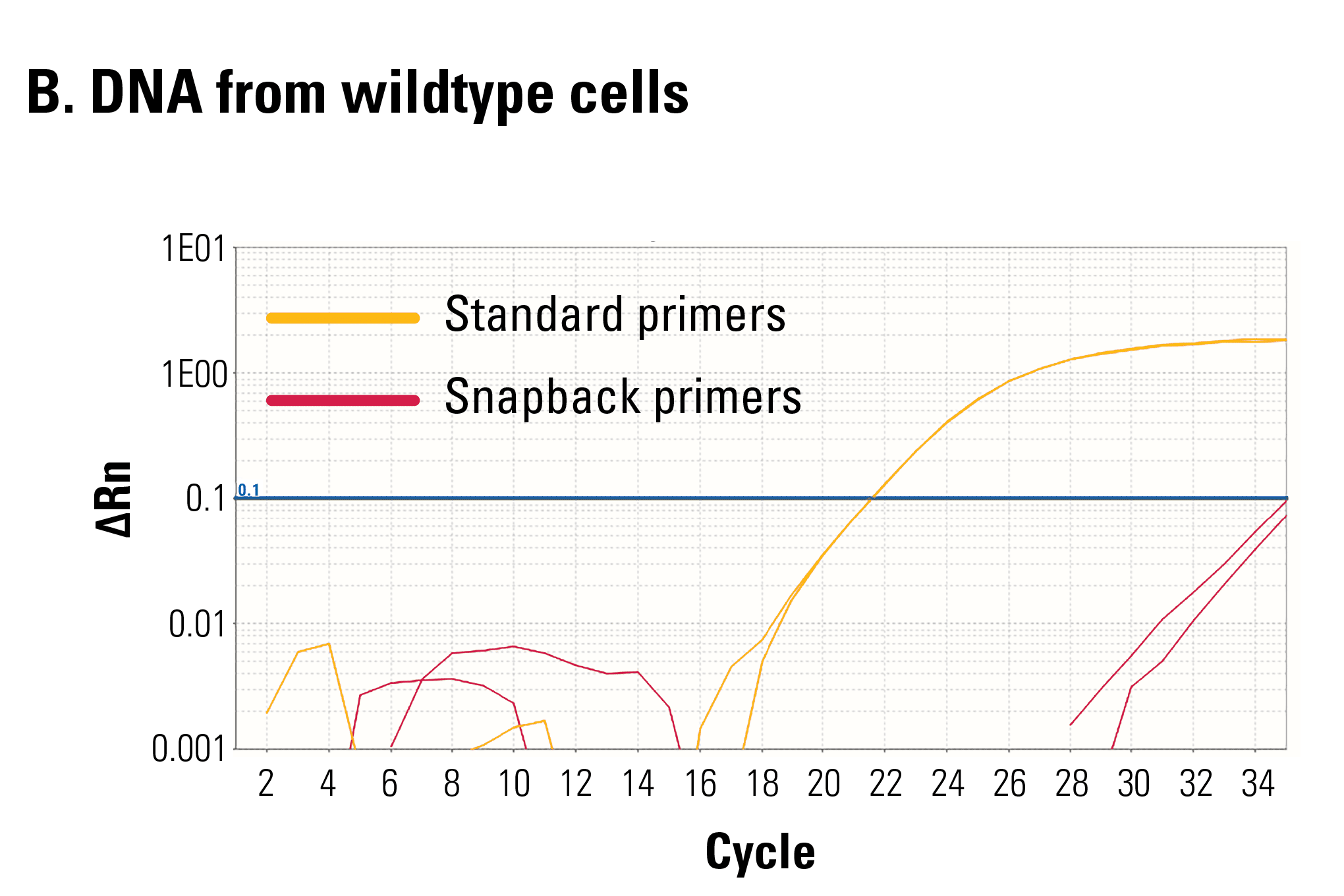
DNA (cytosine-5-)-methyltransferase 3 beta (DNMT3B) 遺伝子を編集したHEK293細胞および未編集のHEK293細胞からDNAを抽出し、本製品を用いて編集効率の測定を行った。標準プライマーでは編集・未編集関わらず、すべてのDNAが増幅される。スナップバックプライマーでは編集済みのDNAのみが増幅されたと考えられる。編集効率は標準プライマーとスナップバックプライマーのCt値差から算出する。
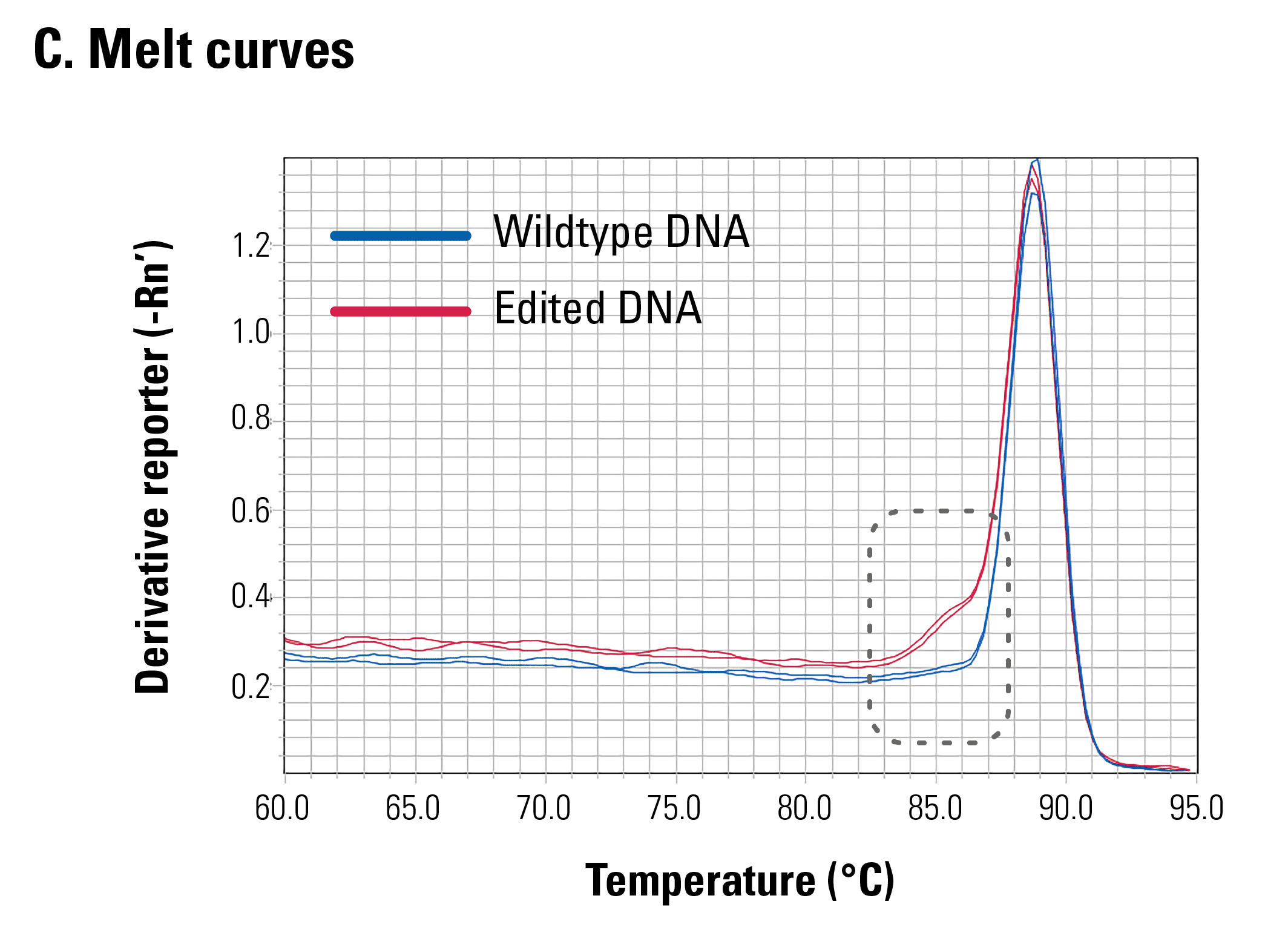
融解曲線の違いも編集済みDNAに欠損があることを示している。
追加しました。
価格
[在庫・価格 :2026年02月22日 00時00分現在]
| 詳細 | 商品名 |
|
文献数 | ||
|---|---|---|---|---|---|
|
CRISPY Master Mix for qPCR-based Gene Editing Quantitation, 200 rxn |
|
本製品は取扱中止になりました | 0 | ||
[在庫・価格 :2026年02月22日 00時00分現在]
CRISPY Master Mix for qPCR-based Gene Editing Quantitation, 200 rxn
文献数: 0
- 商品コード:CRISPY100A-1
- メーカー:SBI
- 包装:1ml
- 本製品は取扱中止になりました
追加しました。
製品情報は掲載時点のものですが、価格表内の価格については随時最新のものに更新されます。お問い合わせいただくタイミングにより製品情報・価格などは変更されている場合があります。
表示価格に、消費税等は含まれていません。一部価格が予告なく変更される場合がありますので、あらかじめご了承下さい。