HOME
>
特集
>
ユビキチン・プロテアソーム特集
HOME
>
試薬
>
細胞情報伝達
>
ユビキチン化・SUMO化
>
ユビキチン化・SUMO化
>
ユビキチン・プロテアソーム特集
HOME
>
試薬
>
細胞情報伝達
>
ユビキチン化・SUMO化
>
プロテアソーム
>
ユビキチン・プロテアソーム特集
Ubiquitin & Proteasome ユビキチン・プロテアソーム特集
掲載日情報:2022/05/19 現在Webページ番号:67461
タンパク質分解、シグナル伝達、炎症応答、DNA損傷修復などの生体内プロセスに関与するユビキチン(Ubiquitin)、類似分子のSUMO、ユビキチンの修飾・脱修飾に関与する各種酵素、ユビキチン化タンパク質の分解を行うプロテアソーム(Proteasome)の研究に有用な製品をまとめてご紹介いたします。
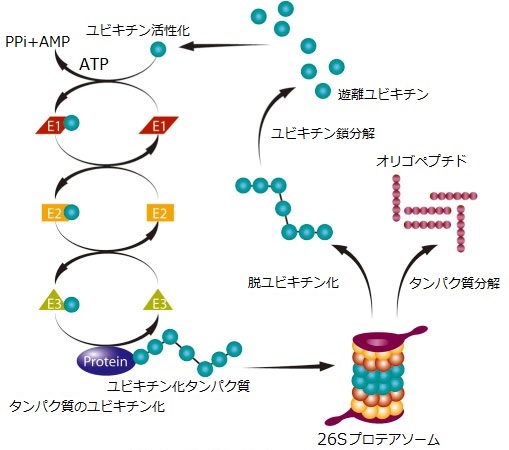
ユビキチンサイクルとタンパク質分解
追加しました。
- 目次
- ユビキチン(Ubiquitin)
- ポリユビキチン鎖
- ユビキチン様タンパク質SUMO
- 脱ユビキチン化酵素(DUB)
- E1酵素(ユビキチン活性化酵素)
- E2酵素(ユビキチン結合酵素)
- E3酵素(ユビキチンリガーゼ)
- プロテアソーム(Proteasome)
- 関連製品:PROTACおよび関連製品
目次
- ユビキチン(Ubiquitin)
- ポリユビキチン鎖
- ユビキチン様タンパク質SUMO
- 脱ユビキチン化酵素(DUB)
- E1酵素(ユビキチン活性化酵素)
- E2酵素(ユビキチン結合酵素)
- E3酵素(ユビキチンリガーゼ)
- プロテアソーム(Proteasome)
- PROTACおよび関連製品
追加しました。
ユビキチン(Ubiquitin)
ユビキチンは76アミノ酸残基からなる翻訳後修飾因子で、真核生物のすべての組織において広く発現しています。タンパク質のユビキチン修飾(ユビキチン化)は、プロテアソームによるタンパク質分解、シグナル伝達、炎症応答、DNA損傷修復などの生体内プロセスに関与しています。ユビキチン化反応は3つの酵素(E1酵素、E2酵素、E3酵素)により段階的に触媒されます。
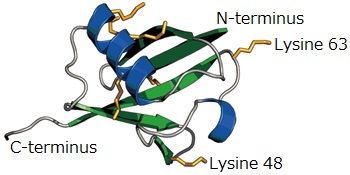

ユビキチンの構造とユビキチン化反応
下記項目をクリックすると製品記事をご覧いただけます。
- ユビキチンの組換え体タンパク質(South Bay Bio社)
- 各種モノユビキチン(R&D Systems社)
- 蛍光標識ユビキチン/蛍光標識ユビキチン様タンパク質(South Bay Bio社)
- ユビキチン化経路関連阻害物質(R&D Systems社)
- ユビキチン化レベル検出用:Proteome Profiler Human Ubiquitin Array Kit(R&D Systems社)
追加しました。
ポリユビキチン鎖
ユビキチンは、単量体(モノユビキチン)および、ユビキチン同士がイソペプチド結合することにより形成されるポリユビキチン鎖の両方の形で存在しています。ポリユビキチン鎖は、1つのユビキチンのC末端グリシン残基(G76)と、もう1つのユビキチンの7種類のリジン残基との間のイソペプチド結合により形成されます。E1酵素、E2酵素、E3酵素、DUBなど、ユビキチン相互作用モチーフを有するタンパク質による、ユビキチンの認識や結合に関するメカニズムの解明に有用です。
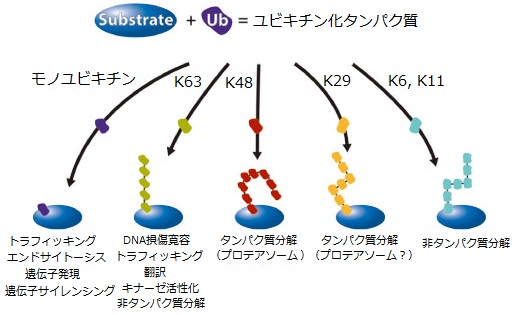
モノユビキチンとポリユビキチン鎖
下記項目をクリックすると製品記事をご覧いただけます。
追加しました。
ユビキチン様タンパク質SUMO
SUMO(small ubiquitin-related modifier)は生物種間で高度に保存されたユビキチン様タンパク質で、翻訳後修飾に関与します。ユビキチンとは一次配列で10%の相同性を有し、ユビキチンに類似した三次構造をとります。タンパク質のSUMO修飾の生理的意義はユビキチン修飾とは異なり、基質タンパク質をタンパク質分解に誘導するのではなく、細胞周期の進行や、核輸送、核における基質タンパク質の局在化、転写制御、タンパク質間相互作用、タンパク質安定性などに関与します。
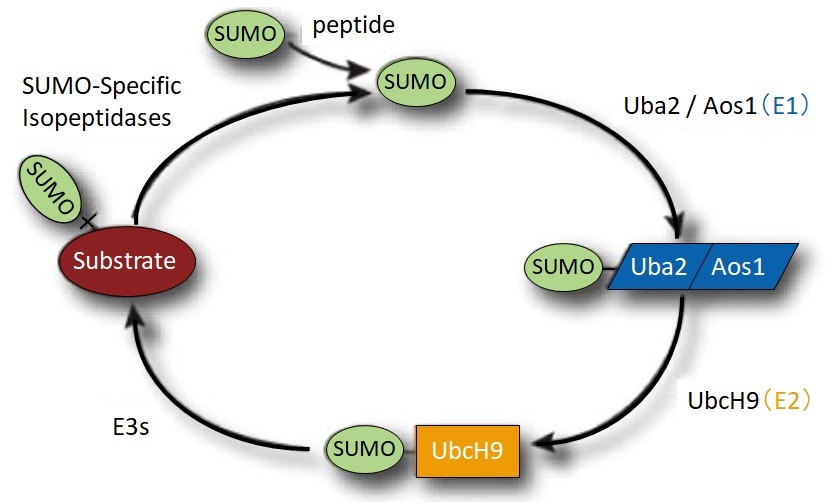
SUMO化と脱SUMO化のサイクル
下記項目をクリックすると製品記事をご覧いただけます。
- ユビキチン様タンパク質SUMO(R&D Systems社)
- Signal-Seeker SUMOylation / Ubiquitination Detection Kit(Cytoskeleton社)
- EpiQuik In Vivo Protein Sumoylation Assay Kit(Epigentek社)
追加しました。
脱ユビキチン化酵素(DUB)
ユビキチン(Ub)やユビキチン様タンパク質(UBL)の修飾・脱修飾は可逆的反応であり、脱修飾反応は特定のシステインプロテアーゼにより触媒されます。システインプロテアーゼは、Ub / UBL前駆体を成熟Ub / UBLにプロセシングしたり、Ub / UBLが付加した基質タンパク質からUb / UBLを脱修飾したり、ポリUb / UBL鎖のイソペプチド結合を切断するなどの機能を有します。
下記項目をクリックすると製品記事をご覧いただけます。
- ユビキチン/ユビキチン様タンパク質プロテアーゼ(R&D Systems社)
- 脱ユビキチン化酵素活性測定(DUB Assay)用製品(South Bay Bio社)
- Cayman DUB Activity Assay Kit(Cayman Chemical社)
- SensoLyte Deubiquitination Assay Kit(AnaSpec社)
追加しました。
E1酵素(ユビキチン活性化酵素)
E1酵素(ユビキチン活性化酵素、E1 Ubiquitin / Ubl Activating Enzyme)はATP依存的にユビキチンC末端をアデニル化し、チオエステル結合を形成します。続いてE1酵素はE2酵素(ユビキチン結合酵素)上のシステイン残基にユビキチンを移します。
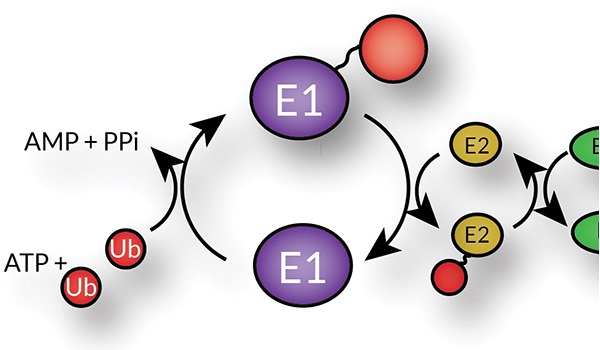
E1酵素(ユビキチン活性化酵素)
下記項目をクリックすると製品記事をご覧いただけます。
追加しました。
E2酵素(ユビキチン結合酵素)
ユビキチンを結合したE2酵素(ユビキチン結合酵素)は、続いて基質タンパク質を認識するE3酵素(ユビキチンリガーゼ)と結合し、ユビキチンを基質タンパク質へ移します。ヒトにおいてE2酵素は約40種類存在します。
[参考文献:PMID 27002219]
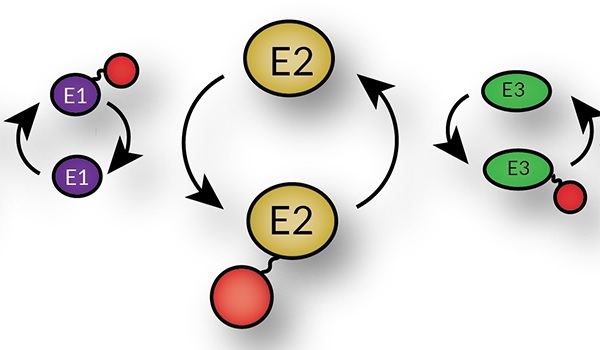
E2酵素(ユビキチン結合酵素E2)
下記項目をクリックすると製品記事をご覧いただけます。
追加しました。
E3酵素(ユビキチンリガーゼ)
E3酵素(ユビキチンリガーゼE3、E3 Ubiquitin Ligase)は数千種類存在し、基質タンパク質に対して非常に高い選択性を有し、それによってユビキチン/ユビキチン様タンパク質が基質タンパク質へ付加される様式が変わります。E3酵素はHECT型とRING型の2つのクラスに大別されます。
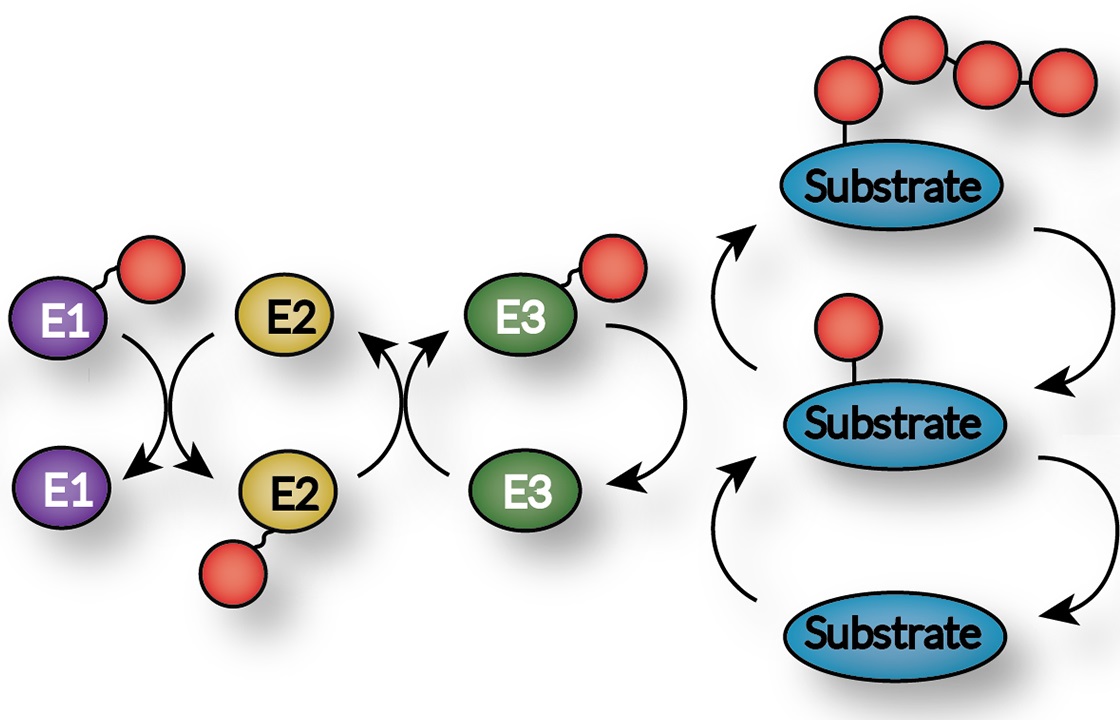
E3酵素(ユビキチンリガーゼ)
- HECT型E3酵素は、触媒システイン残基を含む350残基ほどの大きなドメインを有しており、同システイン残基を介して、E2酵素から基質タンパク質に直接ユビキチンを移行させます。
- RING型E3酵素は触媒活性サイトを有しません。その代わり、RING型E3酵素は巨大な結合表面を有しており、E2酵素と基質タンパク質を近接させるための足場タンパク質として機能します。
下記項目をクリックすると製品記事をご覧いただけます。
- ヒト組換え体E3酵素/E3酵素阻害物質(R&D Systems社)
- ユビキチン関連酵素(BPS Biosciences社)
- 自己ユビキチン化測定キット:E3 Ligase TR-FRET Ubiquitination Kit(South Bay Bio社)
- 自己ユビキチン化測定キット:CBL TR-FRET Assay Kit(BPS Biosciences社)
追加しました。
プロテアソーム(Proteasome)
プロテアソーム(Proteasome)は、真核細胞に広く保存されているタンパク質で、タンパク質分解において機能します。ポリユビキチン鎖が付加された基質タンパク質は、プロテアソームによって認識され、分解されます。ポリユビキチン鎖が、脱ユビキチン化酵素による触媒を受けるとモノマーユビキチンが生成され、ユビキチンがリサイクルされます。
26S Proteasomeは、 プロテアーゼ活性を有する20S Proteasomeと、制御因子19Sから構成されています。20S Proteasomeは、4つのリング構造から成り、1つのリングが7つのサブユニットから構成されています。
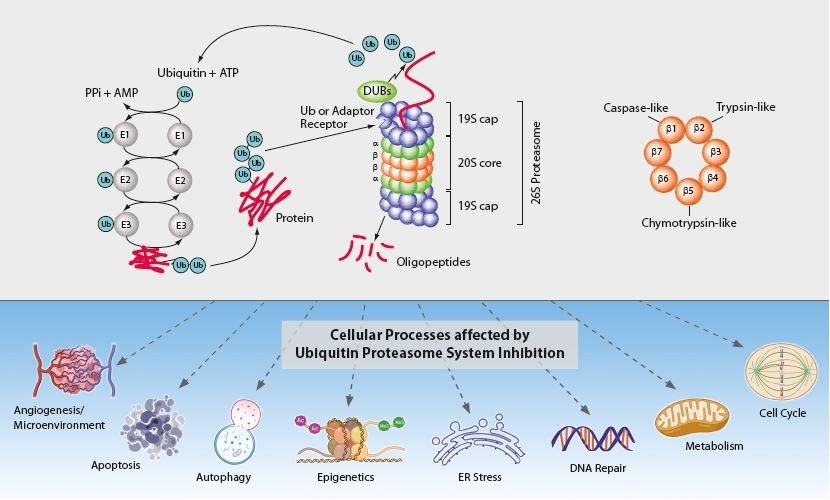
ユビキチン・プロテアソームシステム
下記項目をクリックすると製品記事をご覧いただけます。
- プロテアソーム/プロテアソーム基質(South Bay Bio社)
- プロテアソーム/プロテアソーム阻害物質/基質ペプチド(R&D Systems社)
- 20S proteasome阻害物質と蛍光基質(Adipogen Life Sciences社)
- 20S Proteasome Assay Kit(Cayman Chemical社)
- 20S Proteasome / 20S Immunoproteasome Kit(South Bay Bio社)
追加しました。
関連製品:PROTACおよび関連製品
PROTAC(Proteolysis Targeting Chimera:標的タンパク質分解誘導化合物)は、ユビキチン・プロテアソームシステム(Ubiquitin Proteasome System:UPS)を利用して、細胞内の標的タンパク質の分解(Targeted Protein Degradation, TPD)を誘導する低分子化合物です。
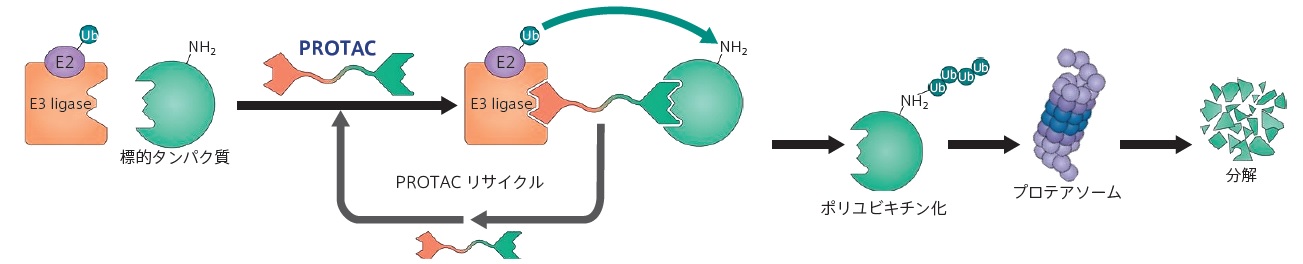
PROTACの触媒作用の模式図
PROTAC化合物は、E3ユビキチンリガーゼ(上図オレンジ色)と標的タンパク質(上図緑色)とを結びつけ三元複合体(ternary complex, TC)を形成する。標的がポリユビキチン化され、プロテアソームによる分解を受ける。PROTACは、ポリユビキチン化反応後TCから離脱し、新たな標的とE3リガーゼとの結合を繰り返すことにより触媒的に作用する。
下記項目をクリックすると製品記事をご覧いただけます。
- PROTAC(標的タンパク質分解誘導化合物)(R&D Systems社)
- PROTAC関連製品(Cayman Chemical社)
- PROTAC阻害物質測定キット(BPS Bioscience社)
- TRIM21経路を利用して標的タンパク質のプロテアソーム分解を行うキット(System Biosciences社)
追加しました。
製品情報は掲載時点のものですが、価格表内の価格については随時最新のものに更新されます。お問い合わせいただくタイミングにより製品情報・価格などは変更されている場合があります。
表示価格に、消費税等は含まれていません。一部価格が予告なく変更される場合がありますので、あらかじめご了承下さい。

