RNA二次構造をハイスループットに予測できます eSHAPE RNA 二次構造解析受託サービス
掲載日情報:2023/12/26 現在Webページ番号:70225
Selective 2'-Hydroxyl Acylation analyzed by Primer Extension(SHAPE)法によって、in vitro転写されたRNAの二次構造の解析を行う受託サービスです。
追加しました。
SHAPE法とは
NAIはRNAに含まれる各塩基の2'-OH基と反応し付加体を形成する性質を持ち、RNA鎖の構造が緩んでいる(塩基対を形成せず、ループ構造または一本鎖となっている)部分においては一部の塩基がNAIと反応します。一方で、RNA二次構造中で塩基対を形成している部分には反応しません。NAIと反応した塩基には逆転写によって変異が誘発されるため、cDNAにおける変異の発生率をコントロール処理と比較することで、RNA二次構造中で塩基対を形成している部位を1塩基レベルで同定することができます。
SHAPE法は、NMRや結晶構造解析などの手法と比較すると、よりハイスループットな解析が可能で、かつ予測アルゴリズムよりも高い精度での構造同定が可能です。
動画 Interrogating RNA Structure and Translation Dynamics with SHAPE and Ribosome Profiling
追加しました。
特長
- 1塩基レベルの解像度でRNA二次構造中の塩基対形成、非形成を予測できます。
- 最小限のシークエンス深度で10,000×を超える、極めて深いRNAカバレッジが得られます。
- 高い精度と再現性が得られます。
追加しました。
解析内容
解析の概要
本サービスでは、1 μg以上のin vitro転写RNAをご提供いただき、下記の手順で解析を行います。
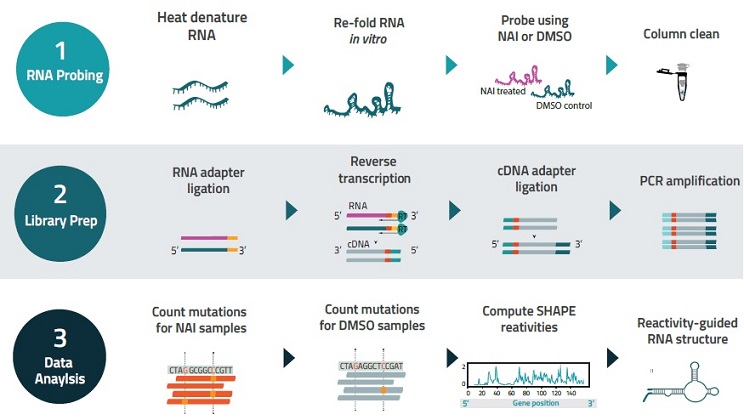
SHAPE Single RNA Kitのワークフロー
- RNAを熱変性し、in vitroでリフォールディングさせる。
- NAIまたはDMSO(ネガティブコントロール)で処理し、クリーンアップ処理する。
- アダプター配列を付加し、逆転写によってcDNAを合成する。
- 合成したcDNAにさらにアダプター配列を付加する。
- PCRで増幅し、ライブラリーを調製する。
- 調製したライブラリーをシークエンシングする。NAI処理試料とDMSO処理試料の間でシークエンシング結果を比較し、標的RNAの各ポジションにおける変異の発生率をプロットする。
納品データ
解析後、下記のデータを納品いたします。
| データの種類 | フォーマット | 概要 |
|---|---|---|
| シークエンス生データ | FASTQ | シークエンサーから直接読み込んで、試料ごとに単離したデータです。 |
| RNAアラインメントデータ | BAM | 下流の追加解析のためにPCR duplicateを除去し、目的のRNAにアラインメントされたリードです。 |
| 変異の発生率 | TSV | NAI処理した試料とDMSO処理(コントロール処理)した試料のRNA塩基ごとの変異の発生率のデータです。 |
| eSHAPE反応性 | BEDGRAPH SHAPE |
1,000倍超のカバレッジが得られた目的RNAの各位置におけるIQR正規化済み反応性スコアです。IGVのようなゲノムビューアで可視化する、またはフォールディング予測をガイドするためのRNA構造解析への入力データとして使用するといった用途向けになります。 |
| eSHAPEサマリーレポート | HTML | eSHAPE解析の結果をインタラクティブな表とプロットでまとめたレポートです。目的RNAのQC metrics、カバレッジ、変異の発生率、反応性データが含まれます。 |
| 反応性比較レポート | HTML | 複数の条件またはRNAを比較した場合の反応性差のレポートです。インタラクティブなプロットと、有意差がみられる部位をp値とともに示した結果表を含みます。 |
追加しました。
解析例
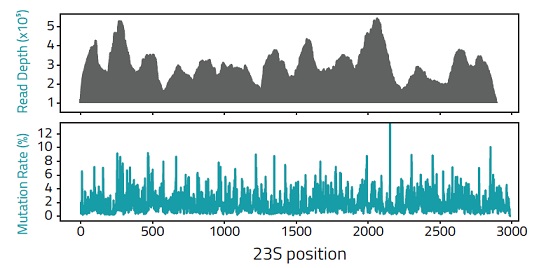
大腸菌の23S rRNA(in vitro転写物)を用いた解析例
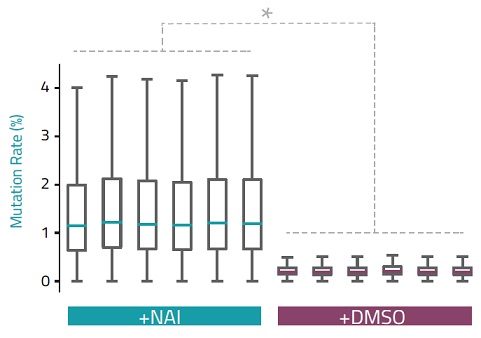
in vitro転写した23S rRNA 100 ngを用いて解析を行った。シークエンシングはNAI処理およびDMSO処理したコントロールの両方から6回分の試料をそれぞれリード数約200万で行った。得られたリードは23S rRNA配列にアラインメントし、配列全体について変異の発生率を計算した。
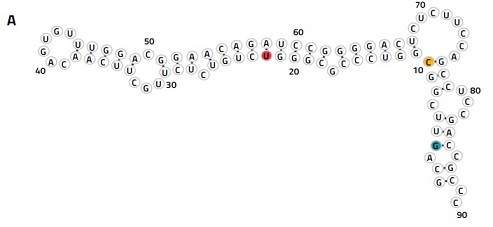
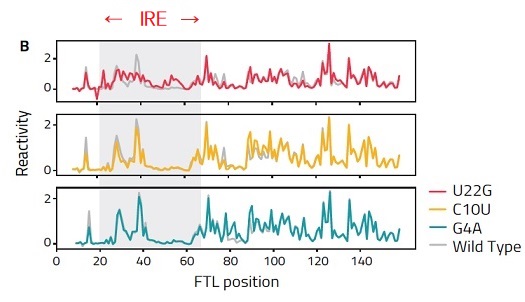
フェリチン軽鎖の解析例
FTL(フェリチン軽鎖)の5'UTRには、安定した鉄応答エレメント(IRE)構造が存在するが、変異によってIRE構造が破壊されると、疾病につながることが知られている。ここでは、in vitro転写mRNAを用いてFTLの5'UTRに3種類の変異型を作製し、NAIとの反応性を検証した。
追加しました。
参考資料
追加しました。
関連した特集記事
追加しました。
ご注文方法/価格
詳細は下記、当社受託・特注品担当までお問い合わせ下さい。
追加しました。
製品情報は掲載時点のものですが、価格表内の価格については随時最新のものに更新されます。お問い合わせいただくタイミングにより製品情報・価格などは変更されている場合があります。
表示価格に、消費税等は含まれていません。一部価格が予告なく変更される場合がありますので、あらかじめご了承下さい。