HOME
>
試薬
>
細胞情報伝達
>
エピジェネティクス
>
Methylamp Methylated DNA Capture Kit <MeDIP Kit>
HOME
>
試薬
>
細胞情報伝達
>
エピジェネティクス
>
DNAメチル化
>
Methylamp Methylated DNA Capture Kit <MeDIP Kit>
HOME
>
試薬
>
遺伝子工学
>
エピジェネティクス
>
Methylamp Methylated DNA Capture Kit <MeDIP Kit>
HOME
>
試薬
>
遺伝子工学
>
エピジェネティクス
>
DNAメチル化
>
Methylamp Methylated DNA Capture Kit <MeDIP Kit>
メチル化DNA濃縮キット(MeDIPキット) Methylamp Methylated DNA Capture Kit <MeDIP Kit>
掲載日情報:2020/06/12 現在Webページ番号:2226
抗メチル化シトシン抗体を用いて、精製したゲノムDNAから迅速かつ効率的にメチル化DNAを濃縮するキットです。
 |
 |
追加しました。
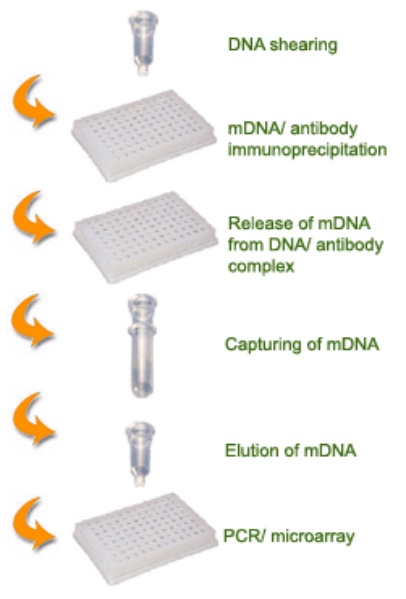
メチル化DNA免疫沈降法(MeDIP)とは
MeDIP is a genome-wide, large scale technique to enrich methylated DNA by isolating methylated DNA fragments with a 5-methylcytosine antibody. This technique has played an important role in helping to identify abnormally methylated genes in diseases such as cancer. With MeDIP, the immunoprecipitated fractions of methylated DNA can be determined by PCR to evaluate the methylation state of individual regions. Additionally, the purified fractions can be input to high throughput DNA detection methods such as DNA microarrays (MeDIP-chip) and next-generation sequencing (MeDIP-seq), becoming a useful approach for methylome-level analysis and for developing comprehensive profiles of DNA methylation on a genome-wide scale.
追加しました。
特長
- 98 %を超える効率でメチル化DNAを濃縮できます。
- ストリッププレートを採用しているため、少ない試料数からハイスループット解析まで、幅広く対応します。
- 操作は3 時間以内で終了します。
- 得られたDNAは定量的・定性的PCR、サザンブロッティング、マイクロアレイ等、様々なDNA検出法に適合します。
- 再現性の良い結果が得られます。
- 適用動物種:Human、Mouse、Ratなど
追加しました。
使用例
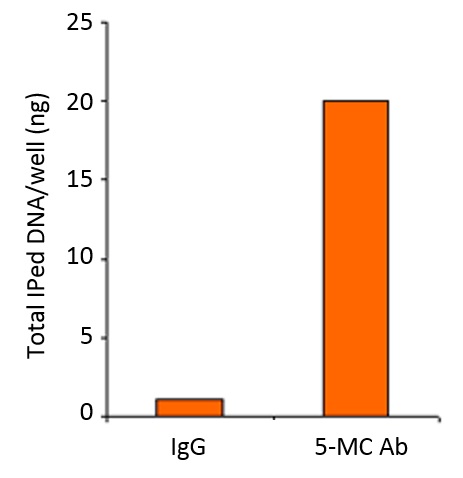
For enrichment of methylated DNA using the kit, DNA (0.5 ug) isolated from MCF-7 cells was added into the microwell. Methylated DNA was captured by 5-mC antibody prebound to the microwells.
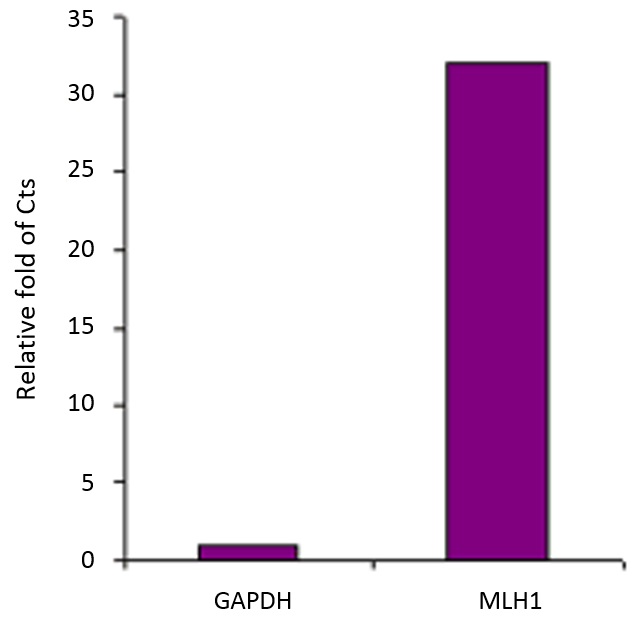
Captured methylated DNA was used for analyzing methylation level of GAPDH and MLH1 promoter with the use of primers and probes specific to GAPDH and MLH1 promoters, respectively.
追加しました。
キット内容
- Antibody buffer
- Reaction buffer
- Wash buffer
- DNA release buffer
- Binding buffer
- Elution buffer
- Normal mouse IgG
- Anti - 5 - methylcytosine
- Proteinase K
- 8-well assay strip
- 8-well strip cap
- F-spin column
- F-collection tube
追加しました。
価格
[在庫・価格 :2026年02月25日 00時01分現在]
| 詳細 | 商品名 |
|
文献数 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Methylated DNA Capture Kit, Methylamp(24samples) |
|
40 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Methylated DNA Capture Kit, Methylamp(48samples) |
|
40 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
[在庫・価格 :2026年02月25日 00時01分現在]
Methylated DNA Capture Kit, Methylamp(24samples)
文献数: 40
- 商品コード:P-1015-24
- メーカー:EPG
- 包装:1kit
- 価格:¥101,000
- 在庫:無(未発注)
- 納期:10日程度 ※※ 表示されている納期は弊社に在庫がなく、取り寄せた場合の目安納期となります。
- 法規制等:
| 説明文 | DNAを試料として,3時間以内にメチル化DNAを濃縮するキット。 |
||||||
|---|---|---|---|---|---|---|---|
| 別包装品 | 別包装品あり | ||||||
| 法規制等 | |||||||
| 保存条件 | 室温,4℃ | 法規備考 | |||||
| 掲載カタログ |
|
||||||
| 製品記事 | |||||||
| 関連記事 | |||||||
追加しました。
使用文献
| Xu X et. al. (2016). Hypoxia-induced Endothelial-Mesenchymal Transition is associated with RASAL1 promoter hypermethylation in human coronary endothelial cells. FEBS Lett. |
| Li W et. al. (2016). Epigenetics reactivation of Nrf2 in Prostate TRAMP C1 Cells by curcumin analog FN1. Chem Res Toxicol. |
| Tan X et. al. (2016). DNMT1 and HDAC2 Cooperate to Facilitate Aberrant Promoter Methylation in Inorganic Phosphate-Induced Endothelial-Mesenchymal Transition. PLoS One. 11(1):e0147816. |
| Tan X et. al. (2016). High inorganic phosphate causes DNMT1 phosphorylation and subsequent fibrotic fibroblast activation. Biochem Biophys Res Commun. |
| Mishra M et. al. (2015). Epigenetic Modification of Mitochondrial DNA in the Development of Diabetic Retinopathy. Invest Ophthalmol Vis Sci. 56(9):5133-42. |
| Guo Y et. al. (2015). Curcumin inhibits anchorage-independent growth of HT29 human colon cancer cells by targeting epigenetic restoration of the tumor suppressor gene DLEC1. Biochem Pharmacol. 94(2):69-78. |
| Hong S et. al. (2015). Epigenetic regulation of genes that modulate chronic stress-induced visceral pain in the peripheral nervous system. Gastroenterology. 148(1):148-157.e7. |
| Tampe B et. al. (2014). Induction of Tet3-dependent Epigenetic Remodeling by Low-dose Hy. EBioMedicine. 1(1) |
| Green TJ et. al. (2014). Anti-viral gene induction is absent upon secondary challenge with double-stranded RNA in the Pacific oyster, Crassostrea gigas. Fish Shellfish Immunol. 39(2):492-7. |
| Skowronki K et. al. (2014). Genome-Wide Analysis in Human Colorectal Cancer Cells Reveals Ischemia-Mediated Expression of Motility Genes via DNA Hypomethylation. PLoS One. 9(7):e103243. |
| Wang J et. al. (2014). MicroRNA-152 Regulates DNA Methyltransferase 1 and Is Involved in the Development and Lactation of Mammary Glands in Dairy Cows. PLoS One. 9(7):e101358. |
| Michailidi C et. al. (2014). Genome-wide and gene-specific epigenomic platforms for hepatocellular carcinoma biomarker development trials. Gastroenterol Res Pract. 2014:597164. |
| Wang HD et. al. (2014). DNA methylation study of fetus genome through a genome-wide analysis. BMC Med Genomics. 7:18. |
| Koch R et. al. (2014). Populational equilibrium through exosome-mediated Wnt signaling in tumor progression of diffuse large B-cell lymphoma. Blood. 123(14):2189-98. |
| Pol Bodetto S et. al. (2013). Cocaine represses protein phosphatase-1Cβ through DNA methylation and Methyl-CpG Binding Protein-2 recruitment in adult rat brain. Neuropharmacology. 73:31-40. |
| Zhang TY et. al. (2013). Epigenetic mechanisms for the early environmental regulation of hippocampal glucocorticoid receptor gene expression in rodents and humans. Neuropsychopharmacology. 38(1):111-23. |
| Tricker PJ et. al. (2012). Low relative humidity triggers RNA-directed de novo DNA methylation and suppression of genes controlling stomatal development. J Exp Bot. 63(10):3799-813. |
| Yu F et. al. (2012). MicroRNA 34c gene down-regulation via DNA methylation promotes self-renewal and epithelial-mesenchymal transition in breast tumor-initiating cells. J Biol Chem. 287(1):465-73. |
| Korostowski L et. al. (2011). Enhancer-driven chromatin interactions during development promote escape from silencing by a long non-coding RNA. Epigenetics Chromatin. 4:21. |
| Zhou FC et. al. (2011). Alcohol alters DNA methylation patterns and inhibits neural stem cell differentiation. Alcohol Clin Exp Res. 35(4):735-46. |
| Hicks SD et. al. (2010). Ethanol-induced methylation of cell cycle genes in neural stem cells. J Neurochem. 114(6):1767-80. |
追加しました。
製品情報は掲載時点のものですが、価格表内の価格については随時最新のものに更新されます。お問い合わせいただくタイミングにより製品情報・価格などは変更されている場合があります。
表示価格に、消費税等は含まれていません。一部価格が予告なく変更される場合がありますので、あらかじめご了承下さい。





